Malgré des ressources nutritives globalement limitées, la surface des océans est peuplée d’une diversité incroyable de microorganismes pourtant a priori en compétition. C’est le paradoxe du plancton

Les microbes marins interagissent constamment entre eux et avec leur environnement, formant des réseaux complexes et dynamiques. Les interactions de ces communautés jouent un rôle écologique crucial pour notre planète.
Ils soutiennent la fixation de dioxyde de carbone et la production d’oxygène via la photosynthèse, en formant la base de la chaîne alimentaire marine, et aussi en régulant le climat. Il est fondamental d’approfondir notre compréhension des interactions trophiques microbiennes étant donné leur capacité à moduler les cycles biogéochimiques, la diversification, et le climat.
La plupart de ces microbes étant difficiles à isoler et à cultiver en laboratoire, la recherche commence à peine à saisir la complexité et la diversité des interactions microbiennes et des interactions symbiotiques qui existent dans la nature. Les récentes campagnes océanographiques à grande échelle des écosystèmes marins (Tara Océans, par exemple) ont récolté des données de génomique environnementale (ou métagénomique) massives qui permettent de reconstruire la diversité génomique du microbiome marin.
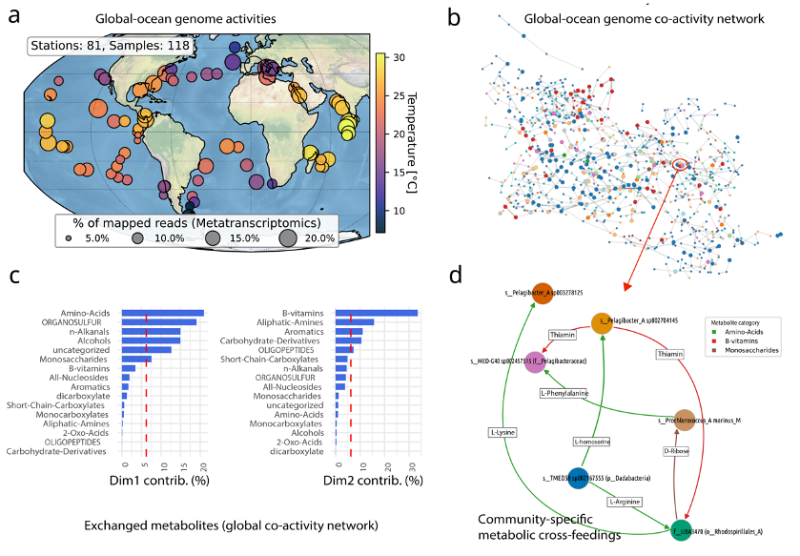
Dans le but d’identifier les consortia de plancton marin à l’échelle mondiale, et de révéler les mécanismes moléculaires influençant leur auto-organisation, une équipe de chercheurs duLS2Net de l’IBENS (CNRS, École Normale Supérieure, Inserm, Université PSL), dirigée par Samuel Chaffron, chargé de recherche CNRS au LS2N, a développé une nouvelle modélisation intégrant information écologique et métabolique (voir illustration ci-dessous) afin de prédire les interactions trophiques potentielles au sein de ces consortia. Grâce aux données Tara Océans, l’intégration d’information écologique sous la forme d’un graphe de génomes co-occurrents globalement, avec des simulations métaboliques des communautés identifiées, a permis de révéler des interdépendances métaboliques principalement médiées par des échanges de certaines vitamines B et d’acides aminés particuliers. Ces prédictions, principalement observées parmi des génomes de petite taille, soulignent leur importance comme mécanisme évolutif global façonnant l’assemblage des communautés de plancton dans l’océan mondial.
Le cadre de modélisation écologique et métabolique développé ici a révélé les fondements génomiques des interdépendances métaboliques prédites qui déterminent l’activité et l’assemblage des communautés de plancton dans l’océan. Il a aussi permis de prédire des interactions métaboliques trophiques entre les cellules les plus abondantes dans l’océan (par exemple, Prochlorococcus et Pelagibacter). Enfin, le cadre informatique développé ici peut directement être appliqué à l’étude d’autres écosystèmes microbiens, dans lesquels les prédictions mécanistes des interactions biotiques peuvent également servir à concevoir de nouvelles hypothèses biologiques et écologiques sur notre planète en pleine mutation.
Contact
Samuel Chaffron Chargé de recherche CNRS au LS2N. samuel.chaffron@ls2n.fr
